MINION UN SEQUENZIATORE DI NUOVA GENERAZIONE PER IDENTIFICARE XYLELLA FASTIDIOSA E LE SUE SOTTOSPECIE
La diagnosi precoce dei patogeni da quarantena è lo strumento più importante per mantenere il territorio libero da questi pericolosi organismi, permettendo di intercettare materiale infetto prima che possa permettere la diffusione del patogeno. E, fra tutti i patogeni da quarantena, particolare attenzione viene data a Xylella fastidiosa, un batterio dalla complessa biologia capace di sviluppare devastanti epidemie in molte diverse specie vegetali.
Per questo, il primo obiettivo del progetto GARDING è stato lo sviluppo di un metodo per la diagnosi e l’identificazione delle diverse sottospecie di Xylella fastidiosa avvalendosi della tecnologia di sequenziamento tramite nanopori utilizzata dal sequenziatore minION.
Il progetto GARDING, finanziato da Regione Lombardia, vede come partner l’Università degli Studi di Verona (Prof. Massimo Delledonne, Dipartimento di Biotecnologie) e di Milano (Prof. Piero Attilio Bianco, Dipartimento di Scienze Agrarie e Ambientali) e l’azienda Vivai Nord, ed ha come obiettivo lo sviluppo di metodi innovativi per la diagnosi di patogeni da quarantena che possono essere veicolati dal commercio di piante ornamentali, contribuendo ad una maggiore sicurezza per i produttori ed i consumatori. Il progetto GARDING si avvale anche di una stretta collaborazione con il Servizio Fitosanitario della Regione Lombardia, organo competente e di grande esperienza in questa tematica.
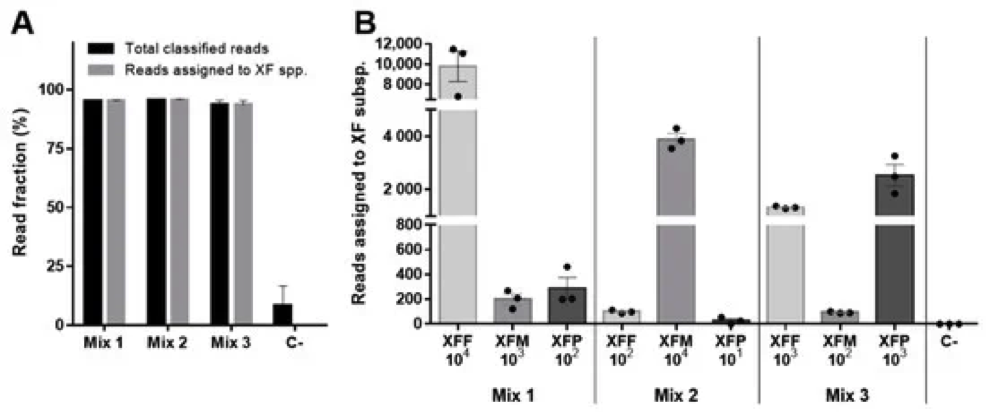
Il primo risultato ottenuto nel progetto è stato lo sviluppo di un metodo diagnostico che permette di ottenere in tempi rapidi una diagnosi accurata, sensibile, e precisa della presenza di Xylella fastidiosa permettendo al contempo di ottenere un’indicazione sulla sottospecie presente nel campione grazie al sequenziamento in tempo reale permesso dall’utilizzo della tecnologia minION. Per raggiungere questo risultato è stata sviluppata una nuova coppia di primer che permettesse l’amplificazione di un gene sufficientemente conservato fra le diverse sottospecie, ma abbastanza diverso da discriminare fra di esse in base ad alcuni polimorfismi di sequenza. Gli ampliconi così ottenuti sono stati poi sequenziati con il minION e, dopo poche decine di minuti, è stato possibile ottenere sequenze in grado di attribuire correttamente gli ampliconi alle diverse sottospecie di Xylella fastidiosa. Questo metodo ha dimostrato di essere efficace lavorando su concentrazioni di patogeno molto basse, anche di dieci volte inferiori rispetto a quanto solitamente rilevato nelle piante infette in natura, fornisce risultati quantitativi, ed è in grado di risolvere anche infezioni miste da parte di diverse sottospecie nello stesso campione.
Questi risultati preliminari aprono delle interessanti prospettive per l’utilizzo di questa tecnologia per la diagnosi di patogeni da quarantena, possibilmente direttamente in campo, essendo altamente portatili.
RIFERIMENTO BIBLIOGRAFICO
Marcolungo, L., Passera, A., Maestri, S., Segala, E., Alfano, M., Gaffuri, F., Marturano, G., Casati, P., Bianco, P.A., Delledonne, M. (2022) Real-time on-site diagnosis of quarantine pathogens in plant tissues by nanopore-based sequencing. Pathogens, 11, 199.

